2019年10月16日, Cell research在线发表了中国科学院分子植物科学卓越创新中心张鹏研究组题为 “Crystal structure of plant PLDα1 reveals catalytic and regulatory mechanisms of eukaryotic phospholipase D”的研究论文。该研究解析了植物磷脂酶Dα1及其与产物磷脂酸(PA)复合体的晶体结构,详细阐释了真核生物磷脂酶D催化磷脂产生磷脂酸及其活性调节的分子机制。
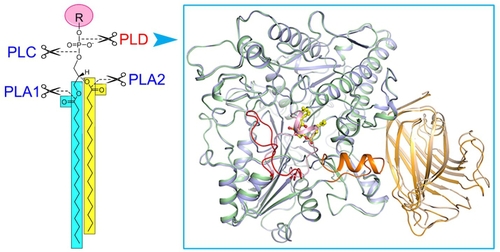
磷脂酶(Phospholipase, PL)催化细胞膜上磷脂的水解。根据水解部分的不同,人们将磷脂酶分成磷脂酶A1、A2、C和磷脂酶D (PLD)(图1)。磷脂酶的功能一方面通过酰基转移实现细胞内磷脂的重新合成与分配;另一方面水解产生脂质信号分子,调控生物体许多重要生理过程,如PLC水解PIP2产生三磷酸肌醇(IP3)和二酰基甘油(DG),PLD水解多种磷脂产生PA。真核生物PLD最先在植物中克隆鉴定,之后在动物及其他生物体中陆续得到鉴定,是生物中高度保守的一类酶。在结构域组成上,PLD包含N端结合磷脂/细胞膜的PX/PH/C2结构域和C端负责催化的HKD结构域。研究发现动物PLD1/2与细胞的生长发育、癌症发生、神经系统疾病如阿尔兹海默症、病毒入侵等疾病的发生密切关联。在植物中,有多达十几种PLD(α1-3、β1-2、γ1-3、δ、ε、ζ1-2)被陆续鉴定。其中ζ1-2与动物中的PLD1/2相似,均属于PX/PH型PLD,其余属于植物特有C2型PLD。植物PLD在体内参与了多种重要生理与病理过程,包括:低温/干旱相应、病原侵染、种子萌发、花粉管伸长、根的发育等。然而,相比于其他三类磷脂酶(A1/A2/C),人们对PLD蛋白的三维结构、催化以及活性调控机制缺乏了解。
在本研究中,我们以高等植物PLD蛋白作为研究对象,成功表达与纯化了有活性的目标蛋白,进而解析了拟南芥PLDα1处于底物非结合态及与产物PA结合态的晶体结构(图1)。这是首次获得真核生物磷脂酶D的三维结构。结构分析揭示了:1)底物的结合口袋是处于两个HKD结构域之间的一个很大的疏水空腔,口袋上方有一个α螺旋和loop组成的盖子,在底物非结合态时,盖子覆盖在口袋上方,处于关闭状态;在产物PA(或底物)结合时,盖子发生构象变化呈开放状态,允许底物进入或者产物结合与释放。2)负责催化底物催化的两个HKD motif呈空间排布且保守,更正了以前文献报道中的错误。3)在活性中心附近存在一个保守的钙离子结合位点,为C2型PLD发挥活性所必须。这一位点在PX/PH型PLD中不存在,很好地解释了为什么C2型PLD需要钙离子激活,而PX/PH型PLD发挥活性不需要钙离子。4)C2结构域与催化结构域的互作网络,C2结构域通过结合质膜/磷脂,从而将PLD定位到细胞膜,同时调节其催化活性。此外,我们的研究还发现已知的动物PLD1/2小分子抑制剂可以抑制PLDα1的活性,表明真核生物PLD蛋白具有相似的结构和催化/抑制机制。这些研究结果使我们对真核生物PLD蛋白的催化和活性调节机制有了全新的认识。研究结果为以真核PLD蛋白为靶标的小分子抑制剂筛选及优化设计指明了方向。
中科院分子植物科学卓越创新中心张鹏研究组副研究员李建戌博士和上海师范大学生命科学学院俞芳副研究员是该论文的共同第一作者,张鹏研究员为本论文的通讯作者。该研究得到了中国科学院(先导B、青促会)、科技部、基金委及上海市的项目资助。实验数据收集工作得到了上海光源19U1/17U1线站的支持与帮助。
数据来源:http://www.sippe.ac.cn/kyjz/kyjz2019/201910/t20191025_5412477.html
图1 脂酶D及晶体结构。
左图显示不同磷脂酶(PLA1、PLA2、PLC、PLD)催化磷脂水解的位点,右图为磷脂酶D与产物磷脂酸(黄色棍棒模型)结合前后的三维结构叠加图,显示底物结合前后的构象变化。